发布时间:2019-04-04 | 来源:统计科学研究室
近日,我所李雷研究团队发表于《生物信息学》期刊的文章“BAUM: improving genome assembly by adaptive unique mapping and local overlap-layout-consensus approach”,经《基因组蛋白质组与生物信息学报》,简称GPB(Genomics, Proteomics and Bioinformatics)评审,入选2018年度“中国生物信息学十大进展”。
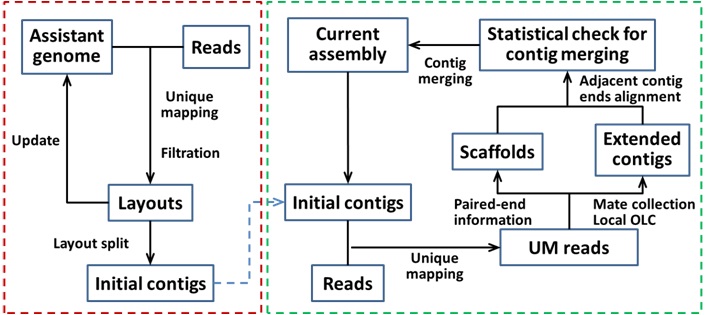
高质量的基因组是进化与遗传研究、精准医疗的基石。基因组拼接是以高通量测序技术为基础的核心计算生物学问题。李雷研究团队将计算数学中的迭代、逐步逼近方法用于基因组拼接,研发了新方法BAUM。BAUM拼接方法的核心是以不同的准则反复映射序列,这可以通过该研究团队原创的SEME映射算法实现。BAUM通过调整映射唯一性准则,量化基因组中由重复序列导致的不确定性。该方法被成功地运用到了高原鼢鼠、鼠兔等基因组的拼接。由于采用逐步逼近方法,BAUM可以成为整合不同测序平台数据的工具。
工具链接:
http://www.zhanyuwang.xin/wordpress/index.php/2017/07/21/baum
原文信息:
Wang A, Wang Z, Li Z, Li LM. BAUM: improving genome assembly by adaptive unique mapping and local overlap-layout-consensus approach. Bioinformatics 2018;34:2019–28. PMID: 29346504
原文链接:
https://academic.oup.com/bioinformatics/article-abstract/34/12/2019/4810438